Resistente Keime: Neues Antibiotikum entdeckt
Die Weltgesundheitsorganisation (WHO) bezeichnet die schleichende und rasant wachsende Zahl antibiotikaresistenter Bakterien als „stille Pandemie“. Diese vielfach hausgemachte Krise wird noch dadurch verschärft, dass in den letzten Jahrzehnten kaum neue Medikamente auf den Markt gekommen sind. Einige Hersteller haben sich komplett aus diesem Bereich zurückgezogen. Schon heute lassen sich deshalb nicht mehr alle Infektionen behandeln und sogar Routine-Eingriffe können zur Gefahr werden.
Dringend neue Wirkstoffe gesucht
Um den Vormarsch antibiotikaresistenter Keime aufzuhalten, braucht es somit dringend neue Wirkstoffe. Eine solche Entdeckung ist nun dem Team um Prof. Dr. Sebastian Hiller vom Biozentrum der Universität Basel und Forscherinnen und Forschern der Northeastern University in Boston gelungen. Die Arbeit entstand im Rahmen des Nationalen Forschungsschwerpunktes (NFS) „AntiResist“.
Suche mittels computerbasiertem Screening
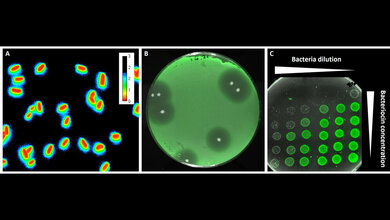
Das neue Antibiotikum Dynobactin haben die Forscherinnen und Forscher durch ein computerbasiertes Screening entdeckt. Es tötet gram-negative Bakterien, zu denen viele gefährliche und resistente Keime wie Klebsiella, E. coli oder Pseudomonas gehören. „Antibiotika gegen diese Gruppe von Bakterien zu finden, ist alles andere als trivial“, sagt Hiller. „Sie sind durch ihre doppelte Membran gut geschützt und bieten daher nur wenig Angriffsfläche. Und in den Millionen Jahren ihrer Evolution haben sie zahlreiche Wege gefunden, Antibiotika unschädlich zu machen.“
Bakterien stellen selbst antibiotisch wirkende Peptide her
Erst im vergangenen Jahr hat Hillers Team das Wirkprinzip des kürzlich entdeckten Peptidantibiotikums Darobactin entschlüsselt. Diese Erkenntnisse flossen direkt in die Suche nach neuen Antibiotika ein. Dabei machten sie sich unter anderem zu Nutze, dass viele Bakterien selbst antibiotisch wirkende Peptide herstellen, um sich gegenseitig zu bekämpfen. Und dass diese Peptide, im Gegensatz zu Naturstoffen, im Erbgut der Bakterien festgeschrieben sind.
„Die Gene für solche Peptidantibiotika besitzen ein klares Erkennungszeichen“, erklärt Ko-Erstautor Dr. Seyed M. Modaresi. „Nach diesem Merkmal hat der Rechner das gesamte Erbgut von Bakterien, die solche Peptide produzieren, systematisch durchforstet. Dabei sind wir auf Dynobactin gestoßen.“ Dass es äußerst wirksam ist, konnten die Autoren in ihrer Studie zeigen. Mäuse mit einer lebensgefährlichen Blutvergiftung durch resistente Bakterien überstanden die schwere Infektion durch die Gabe von Dynobactin.
Dynobactin blockiert Membranprotein BamA
Durch eine Kombination verschiedener Methoden konnten die Forscherinnen und Forscher die Struktur und die Wirkungsweise von Dynobactin ermitteln. Es blockiert das bakterielle Membranprotein BamA, welches beim Aufbau und der Erneuerung der äußeren Schutzhülle der Keime eine wichtige Rolle spielt. „Dynobactin steckt von außen wie ein Korken im BamA und hindert es daran, seine Aufgaben zu erfüllen. Die Bakterien sterben“, so Modaresi. „Obwohl Dynobactin chemisch kaum Ähnlichkeiten mit dem bekannten Darobactin aufweist, bekommt es die Bakterien an derselben Stelle zu fassen. Damit hatten wir anfangs nicht gerechnet.“
Noch mehr Peptide auf ihre Tauglichkeit hin prüfen
Auf molekularer Ebene jedoch, so stellten die Forscherinnen und Forscher fest, interagiert Dynobactin anders mit BamA als Darobactin. Indem man bestimmte Eigenschaften der beiden kombiniert, ließen sich die potenziellen Wirkstoffe weiter verbessern und optimieren. Dies ist ein wichtiger Schritt auf dem Weg zu einem wirksamen Medikament. „Die computerbasierte Screening-Methode wird der Suche nach den dringend benötigten Antibiotika einen neuen Schub verleihen“, ist sich Hiller sicher. „Zukünftig wollen wir das Ganze erweitern und noch mehr Peptide auf ihre Tauglichkeit hin prüfen.“
Quelle: idw/Uni Basel
Artikel teilen