Wenn sich ein Protein falsch zusammenfaltet, kann das teilweise schwerwiegende Folgen haben. Krankheiten wie Parkinson beruhen auf diesen fehlgefalteten Proteinen. Um diese Strukturveränderungen zu erkennen, waren bisher spezielle Verfahren notwendig. Nun entwickelten Forschende der Universität Göttingen ein Verfahren, bei dem mit einfacher Lichtmikroskopie in Kombination mit Künstlicher Intelligenz (KI) 3D-Strukturen dargestellt werden können.
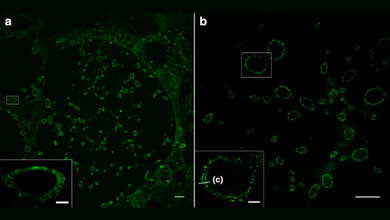
Darstellung mit herkömmlichem Lichtmikroskop
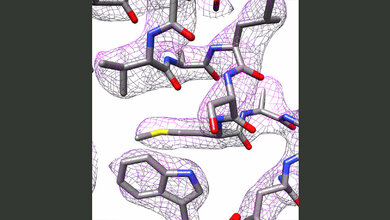
Die Zeitschrift Nature zeichnete die sogenannte ONE-Mikroskopie (One-step Nanoscale Expansion) als eine der „sieben Technologien, die man 2024 im Auge behalten sollte“ aus. Sie wurde von Prof. Dr. Silvio Rizzoli und Dr. Ali Shaib entwickelt. Anstelle teurer und hochauflösender Mikroskope für eine bessere Auflösung verbesserten sie mit der einstufigen Nanoskalen-Expansion die Darstellung im Lichtmikroskop. Das neue Verfahren vergrößert das Volumen der Probe, indem die darin enthaltenen Zellen und Strukturen auf ein wasserabsorbierendes GEl gebunden werden. Es dringt in die Zellen ein. Durch die Aufnahme von Wasser vergrößert sich das Gel bis auf das 15-fache seines Volumens.
.

Dadurch bewegen sich auch die Moleküle der Probe auseinander und werden größer. Nach gezielter Markierung mit fluoreszierenden Molekülen können sie dann im Lichtmikroskop abgebildet werden. Wenn anschließend die KI eingesetzt wird zur Auswertung der Fluoreszenzveränderungen, erhielten die Forschenden Ergebnisse, die bisher nur mit hochauflösenden Kryo-Elektronenmikroskopen oder Röntgentechnologie möglich war.
Open-Source-Paket
Aus zweidimensionalen Fluoreszenzbildern können nun 3D-Proteinstrukturen rekonstruiert werden – was bisher unvorstellbar war. Veränderungen in der räumlichen Struktur können so leicht gefunden werden oder auch Details einzelner Proteine und Multiproteinkomplexen. An Hirnwasser-Proben von Patientinnen und Patienten konnten so bereits die Veränderungen der Proteine typisch für Parkinson festgestellt werden: eine verbesserte Früherkennung, die eine frühe Behandlung ermöglicht.
Die ONE-Mikroskopie ist einfach, kostengünstig und kann in jedem Labor durchgeführt werden. So werden Auflösungen im Bereich von ca. einem Nanometer erreicht. Die Autoren stellen die benötigte Software als kostenloses Open-Source-Paket zur Verfügung.
Quelle: idw
Artikel teilen