Die Forscher kombinierten die hochauflösende Fluoreszenzmikroskopie dSTORM mit der Expansionsmikroskopie ExM. Federführend bei der Publikation war ein Team vom Biozentrum der Julius-Maximilians-Universität (JMU) Würzburg: Professor Markus Sauer, Leiter des Lehrstuhls für Biotechnologie und Biophysik, mit den Doktoranden Fabian Zwettler und Sebastian Reinhard. Maßgeblich beteiligt waren auch die Professoren Paul Guichard von der Universität Genf (Schweiz) und Toby Bell von der Monash University (Australien).
Probleme behoben
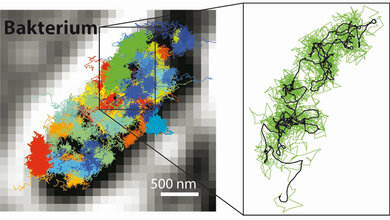
Durch dSTORM ist eine fast molekulare Auflösung von circa 20 Nanometern möglich. Seit Kurzem ist außerdem die Expansionsmikroskopie verfügbar, sodass sich die Forscher durch eine Kombination der Methoden eine noch höhere Auflösung erhofften.
Bei der ExM wird die zu untersuchende Probe in ein quellbares Polymer vernetzt. Dann zerstört man die Wechselwirkungen der Moleküle in der Probe und lässt diese mit Wasser aufquellen. Dadurch kommt es zu einer Expansion: Die abzubildenden Moleküle driften um den Faktor vier räumlich auseinander.
Bisher war eine Kombination der Methoden aus zwei Gründen nicht möglich. Zum einen überlebten die bei dSTORM eingesetzten Fluoreszenzfarbstoffe die Polymerisation des wässrigen Gels nicht. Zum anderen benötigt dSTORM eine Pufferlösung. Durch sie zog sich jedoch die expandierte Probe wieder zusammen.
Fluoreszenzimaging mit molekularer Auflösung
Die Stabilisierung des Gels und die Immunfärbung nach der Expansion führte die Forscher schließlich zum Erfolg. Der Abstandsfehler reduziere sich somit auf nur noch fünf Nanometer, so Sauer. So sei erstmals ein Fluoreszenzimaging mit molekularer Auflösung durchführbar.
Als Beispiel ihrer Methode zeigten die Forscher Tubulinröhrchen als hohe Zylinder mit einem Durchmesser von 25 Nanometern. An den Zentriolen gelang es, Dreiergruppen aus Tubulinstrukturen mit einem Abstand von 15 bis 20 Nanometer scharf abzubilden. Die Zentriolen sind Zellstrukturen, die bei der Zellteilung eine wichtige Rolle spielen. Das Team will darum als nächstes die Methode bei verschiedenen Strukturen, Organellen und Multiproteinkomplexen der Zelle anwenden.
Fabian U. Zwettler, Sebastian Reinhard, Davide Gambarotto, et al.: Molecular resolution imaging by post-labeling expansion single-molecule localization microscopy (Ex-SMLM). Nature Communications, 7. Juli 2020, doi.org/10.1038/s41467-020-17086-8.
Quelle: Universität Würzburg
Artikel teilen