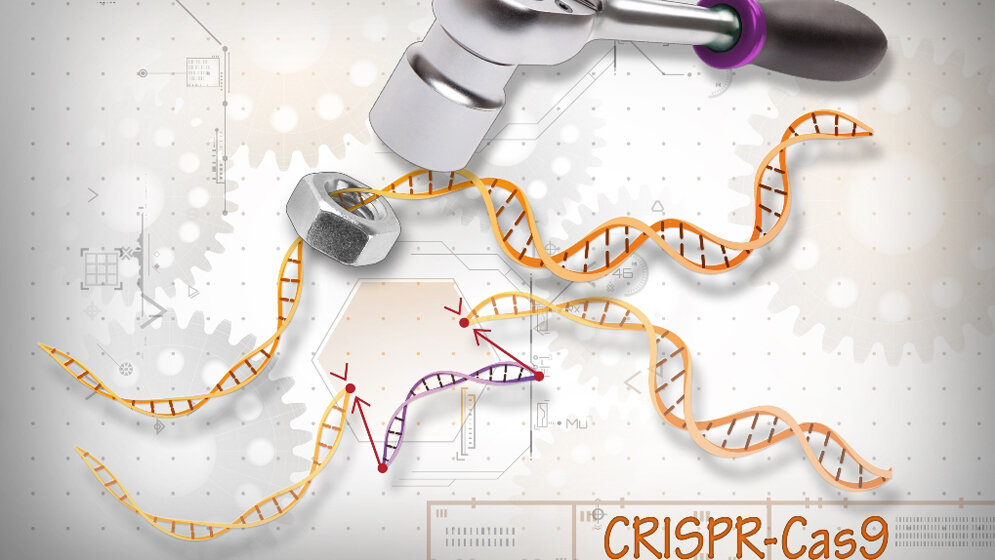
Krebs und viele andere Erkrankungen beruhen auf Gendefekten. Häufig kann der Körper den Ausfall eines Gens kompensieren; erst die Kombination mehrerer genetischer Fehler führt zum Krankheitsbild. Eine Möglichkeit, Millionen solcher Kombinationen von Gendefekten zu simulieren und ihre Auswirkungen in der Zellkultur zu untersuchen, bietet jetzt die an der Goethe-Universität Frankfurt entwickelte 3Cs-Multiplex-Technik auf Basis der CRISPR-Cas-Technologie. CRISPR-Cas ist eine „Genschere“, die es erlaubt, gezielt Gene einzuschleusen, zu entfernen und auszuschalten. Dazu werden kleine Erbgut-Schnipsel („single guide RNA“) als „Adresse“ genutzt, die die Genschere zu bestimmten Abschnitten der DNA leiten, wo die Genschere dann aktiv wird.
Erweiterung der 3Cs-Technik
Zur Entwicklung der 3Cs-Multiplex-Technik erweiterten Wissenschaftlerinnen und Wissenschaftler vom Institut für Biochemie II der Goethe-Universität ihre vor drei Jahren entwickelte und patentierte 3Cs-Technik. 3Cs steht für covalently-closed circular-synthesized, weil die für CRISPR-Cas eingesetzten RNA-Elemente dabei mit Hilfe einer kreisförmigen Synthese generiert somit einheitlicher verteilt sind. Mit einer ganzen Bibliothek solcher RNA-Ringe lässt sich jedes beliebige Gen in einer Zelle gezielt adressieren, um es zu verändern oder auszuschalten.
Gleichzeitige Manipulation zweier Gene
Die neue 3Cs-Multiplex-Technik erlaubt nun sogar die gleichzeitige Manipulation zweier Gene in einer Zelle. Biochemiker Dr. Manuel Kaulich von der Goethe-Universität Frankfurt erläutert: „Wir können ‚Adress‘-RNA-Bibliotheken für alle denkbaren Zweier-Kombinationen von Genen herstellen. Damit lassen sich bis zu mehreren Millionen Kombinationen gleichzeitig in einem Experiment testen.“
Bislang war der Aufwand für solche Experimente sehr hoch; die neue Technik der Arbeitsgruppe reduziert ihn einschließlich der Kosten um den Faktor Zehn. Denn das Team kann die Adress-Bibliotheken dank der neuen 3Cs-Multiplex-Technik sehr einheitlich und qualitativ hochwertig herstellen. „Durch die mäßige Qualität der bislang verfügbaren CRISPR-Cas-Bibliotheken mussten immer sehr große Experimente durchgeführt werden, um entstehende Fehler statistisch auszugleichen“, so Kaulich.
Autophagie-Mutationen untersucht
Am Beispiel von verschiedenen an Abbauprozessen beteiligten Genen zeigte die Arbeitsgruppe das Potenzial der neuen 3Cs-Multiplex-Technik: Sie untersuchte knapp 13.000 Zweierkombinationen von Genen, die für Recyclingprozesse (Autophagie) in der Zelle verantwortlich sind. Mit deren Hilfe baut die Zelle „ausgediente“ Zellbestandteile ab und verwertet sie. Störungen der Autophagie können Zellwucherungen auslösen.
„Mit der 3Cs-Multiplex-Technik konnten wir zum Beispiel zwei an der Autophagie beteiligte Gene identifizieren, deren Ausschalten zu einem unkontrollierten Wachstum von Zellen führt“, erklärt Kaulich. „Genau diese Autophagie-Mutationen kommen bei jedem fünften Patienten mit einem Plattenephithelkarzinom der Lunge vor. Auf diese Weise können wir in Zellkulturexperimenten sehr effizient nach Genen suchen, die bei Krebs oder auch Krankheiten des Nerven- und Immunsystems eine wichtige Rolle spielen und die sich als mögliche Ziele für Therapien eignen.“
Die Arbeitsgruppe der Goethe-Universität hat ihre Entwicklungen über die universitäre Technologietransfer-Tochter Innovectis zum Patent angemeldet. Das aus dem Institut für Biochemie II unter Beteiligung von Manuel Kaulich ausgegründete Start-up-Unternehmen Vivlion GmbH bietet die Nutzung der Technologie bereits auf dem Markt an.
Valentina Diehl, Martin Wegner, Paolo Grumati, et al.: Minimized combinatorial CRISPR screens identify genetic interactions in autophagy. Nucleic Acids Research, gkab309, DOI: doi.org/10.1093/nar/gkab309.
Quelle: idw/Goethe-Universität Frankfurt am Main
Artikel teilen