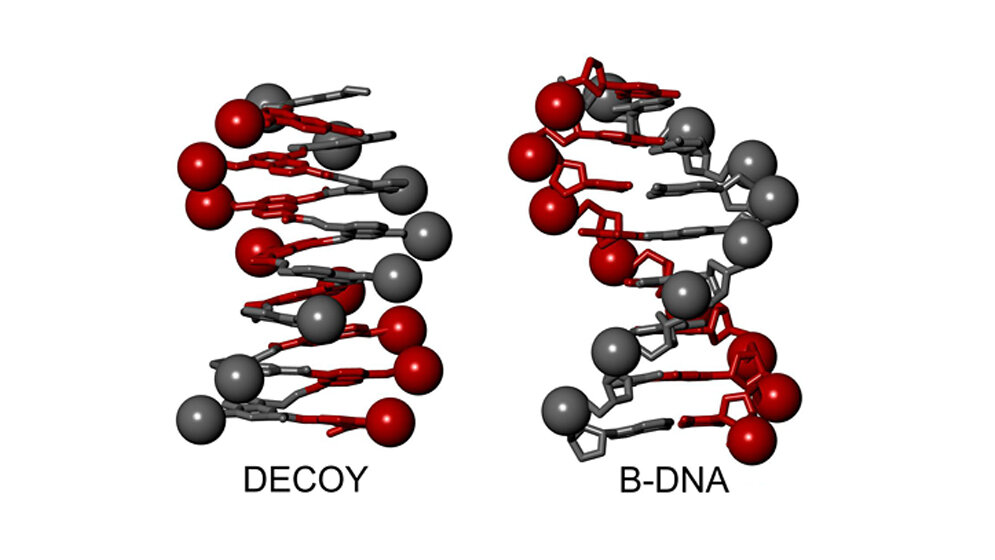
Ivan Huc ahmt in seiner Forschung die Prinzipien der Natur nach, und das auf kleinster Ebene. Der Chemiker schafft mit seiner Arbeitsgruppe „Biomimetic Supramolecular Chemistry“ an der LMU künstliche Moleküle, die sich mithilfe einer Art Origami-Technik nach dem Abbild ihrer natürlichen Vorbilder formen lassen, Foldamere nennt er diese. Nun ist es Ivan Huc gelungen, Eigenschaften der Oberfläche einer DNA-Doppelhelix so nachzuahmen, dass Proteine mit dem Imitat interagieren. Darüber berichtet der Chemiker, der bis Sommer 2017 an der Universität Bordeaux, Frankreich, forschte, aktuell in der Fachzeitschrift Nature Chemistry. In der Studie blockierten die künstlichen DNA-Imitate verschiedene Enzyme von Viren, darunter die HIV Integrase, durch die der HI-Virus sein Genom in die Wirtszelle einschleust. Damit könnte seine Forschung ganz neue therapeutische Ansatzpunkte eröffnen.
Form bestimmt die Funktion
Hucs aktuelle Veröffentlichung baut vor allem auf zwei früheren Arbeiten auf, die ebenfalls in diesem Jahr in Nature Chemistry erschienen sind. Darin zeigte er, durch welche Interaktionsmuster künstliche Moleküle organische Formen wie die Helixstruktur annehmen können und unter welchen Umständen Ribosomen künstlichen Molekülen gegenüber tolerant sind. „Die Form bestimmt die Funktion“, erklärt Ivan Huc seinen Ansatz. In der nun neu erschienenen Studie hat der Chemiker als Basis ein künstliches Molekül entwickelt, das sich schraubenförmig falten und nach einer Art Baukasten-Prinzip vielfach modellieren lässt. So konnte Ivan Huc Oberflächeneigenschaften der natürlichen DNA-Doppelhelix imitieren. Das Imitat ist so gut geworden, dass zwei Enzyme, darunter die HIV Integrase, auf die falsche DNA hereinfallen und dadurch blockiert werden können.
Natur muss quasi „übertroffen“ werden
Damit dies auch dann funktioniert, wenn die Enzyme sowohl die künstliche als auch die echte DNA zur Wahl haben, muss Hucs Schöpfung die Natur quasi übertreffen: „Wenn die Enzyme auch unter konkurrierenden Bedingungen an das Foldamer binden sollen, muss das Imitat besser sein als die DNA selbst“, erläutert Huc. Tatsächlich ist dies in der Studie gelungen: Die Bindung der HIV Integrase an das Foldamer war stärker als an die DNA selbst. „Obwohl das Design auf die Ähnlichkeit zur DNA abzielt, verdankt das Foldamer seine wertvollsten Eigenschaften gerade seinen Unterschieden zur DNA“, betont Huc.
Enzyme blockieren?
Das Baukastenprinzip, nach dem sich die künstlichen DNA-Sequenzen nach Belieben gestalten lassen, eröffnet viele Variationsmöglichkeiten. In der vorliegenden Studie testete Ivan Huc die Funktion am Beispiel von Enzymen, die an eine beliebige Stelle der DNA binden. Es wäre jedoch auch denkbar, künstliche DNA-Abschnitte zu entwickeln, um Enzyme zu blockieren, die nur an bestimmte DNA-Sequenzen binden. (idw, red)
Krzysztof Ziach, Céline Chollet, Vincent Parissi, et al.: Single helically folded aromatic oligoamides that mimic the charge surface of double-stranded B-DNA. Nature Chemistry (2018), DOI:10.1038/s41557-018-0018-7, Published online: 02 April 2018.
Joseph M. Rogers, Sunbum Kwon, Simon J. Dawson, et al.: Ribosomal synthesis and folding of peptide-helical aromatic foldamer hybrids. Nature Chemistry, Volume 10, pages 405–412 (2018), DOI:10.1038/s41557-018-0007-x.
Soumen De, Bo Chi, Thierry Granier, et al.: Designing cooperatively folded abiotic uni- and multimolecular helix bundles. Nature Chemistry volume 10, pages 51–57 (2018), DOI:10.1038/nchem.2854.
Artikel teilen