Wie Coronaviren Zellen umprogrammieren
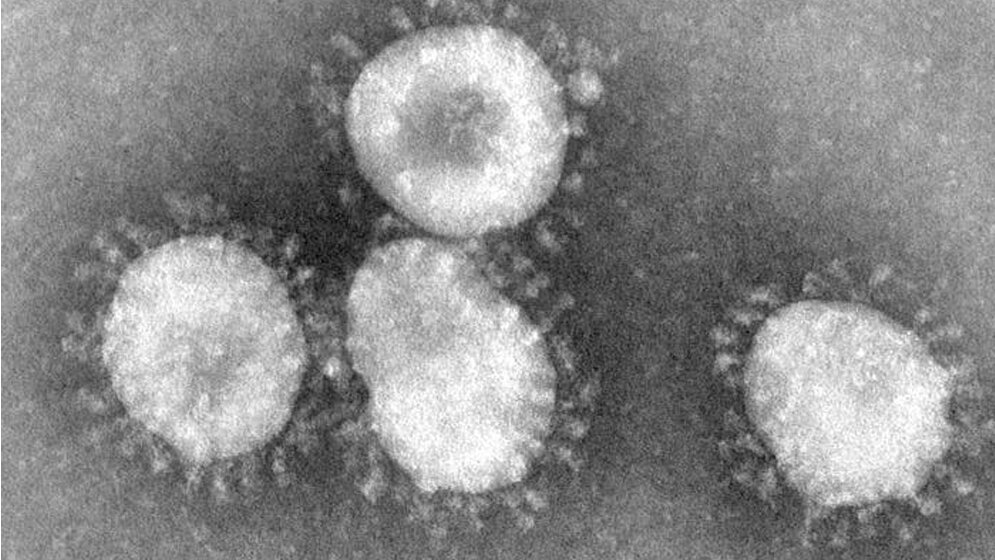
Coronaviren sind weltweit verbreitete wichtige Verursacher von humanen und tierischen Erkrankungen, insbesondere der Atmungsorgane. Durch ihr großes Genom, das größte bekannte Genom aller RNA-Viren, können sie sich besonders vielfältig und schnell an neue Situationen anpassen. Wie bewerkstelligen die Coronaviren es, den zellulären Stoffwechsel so umzuprogrammieren, dass neue infektiöse Viruspartikel produziert werden? Und was sind die molekularen Ursachen der unterschiedlichen Krankheitsverläufe durch verschiedene Coronavirus-Infektionen? Ein interdisziplinäres Forscherteam der Justus-Liebig-Universität Gießen (JLU)hat nun die Genantwort der Wirtszelle und ihre epigenetischen Kontrollmechanismen entschlüsselt.
Coronaviren bekannt von SARS und MERS
Die vier bekannten humanen Coronaviren, zu denen auch das in der Studie untersuchte Coronavirus 229E gehört, verursachen überwiegend relativ milde verlaufende und vorübergehende Infektionen der oberen Luftwege. „Infektionen mit den ähnlich aufgebauten verwandten zoonotischen Coronaviren SARS-CoV und MERS-CoV hingegen können zu schwersten Lungenentzündungen bis hin zum Lungenversagen führen, wenn diese Viren aus dem Tier in einen menschlichen Wirt wechseln“, so Prof. Dr. John Ziebuhr, Virologe an der JLU. Wie alle Viren benötigen auch Coronaviren einen geeigneten Wirt, um sich zu vermehren. Nach dem Eindringen in spezifische Wirtszellen wird das Coronavirus-Genom im Zytoplasma infizierter Zellen freigesetzt und dort vermehrt. In den erkrankten Organen finden sich vermehrt Botenstoffe des angeborenen Immunsystems, insbesondere sogenannte Zytokine, und entzündliche Veränderungen. Basierend auf ihrer Expertise in der Analyse molekularer Entzündungsvorgänge haben Wissenschaftlerinnen und Wissenschaftler aus der Pharmakologie nun in Zusammenarbeit mit Kollegen/-innen aus der Virologie, der Biochemie und der Genetik die molekularen Vorgänge in den infizierten Zellen systematisch erfasst.
Steuerung deutlich komplexerer biologische Funktionen
Bioinformatische Analysen zeigten, dass das virusregulierte Genspektrum vermutlich deutlich komplexere biologische Funktionen steuert, als das einer nur entzündlich aktivierten Zelle. Um besser zu verstehen, wie ein im Zytoplasma replizierendes Virus so umfassend die Genomfunktionen einer Wirtszelle beeinflussen kann, kartierten die Forscherinnen und Forscher fünf epigenetische „Fingerabdrücke“ der DNA-Hüllproteine (den Histonen). „Wir haben über tausend durch Coronaviren aktivierte DNA-Elemente, sogenannte Enhancer, gefunden, die ein eigenes Muster bilden und offenbar dafür sorgen, dass nur ganz bestimmte Gene des Zellstoffwechsels so aktiviert werden, dass sie dem Virus nützen. Gleichzeitig werden andere DNA Bereiche im Zellkern abgeschaltet oder ihre Aktivität gedämpft – offenbar um Genprodukte, die die Zellen schützen oder andere Immunzellen anlocken könnten, zu blockieren“, erklärt Marek Bartkuhn, Genetiker und Bioinformatiker an der JLU. Coronaviren führen also zu einer genomweiten Reprogrammierung von Funktionen im Zellkern.
Coronaviren hemmen NF-κB
In einem weiteren Ansatz unterbrachen die Forscherinnen und Forscher die Signalwege, die zur Aktivierung des Transkriptionsfaktors NF-κB, einem zentralen genetischen Schalter von Immunvorgängen, führen und untersuchten die Konsequenzen sowohl für die Virusreplikation als auch für die Wirtszellfunktionen. So konnten sie zeigen, dass Coronaviren die Aktivität dieses wichtigen Faktors deutlich hemmen – wodurch eine mögliche Abwehrreaktion der Wirtszelle abgeschwächt wird –, aber nicht komplett aufheben. Dadurch bleiben bestimmte Zellfunktionen noch erhalten, die das Virus offenbar braucht.
Wege zum Hemmen Coronavirus-spezifischer Gene
„Diese Daten sind ein sehr interessantes Beispiel dafür, wie clever Mikroben die Balance von intrazellulären Signalwegen beeinflussen, um sich einen Vorteil zu verschaffen“, sagt Prof. Dr. Lienhard Schmitz vom Biochemischen Institut der JLU, der schon seit Jahren zusammen mit dem Leiter der Studie, dem Pharmakologen Prof. Dr. Michael Kracht vom Rudolf-Buchheim-Institut für Pharmakologie der JLU, das NF-κB-System molekular untersucht. „Wir haben im Rahmen dieser Untersuchungen einerseits besser verstanden, wie ein Coronavirus mechanistisch funktioniert“, so Prof. Kracht. „Zum anderen haben wir mit Hilfe von pharmakologischen Substanzen und neuen genetischen Methoden wie der RNA-Interferenz und der Genschere Crispr/Cas9 auch Wege gefunden, die Coronavirus-spezifischen Gene gezielt zu hemmen.“
Ansätze weiterentwickeln
Diese Ansätze sollen nun im Rahmen der neuen klinischen Forschergruppe (KFO 309 „Virus-induziertes Lungenversagen – Pathobiologie und neue Therapiestrategien“), die sich mit Virusinfektionen der Lunge beschäftigt, in krankheitsnäheren Situationen weiterentwickelt werden. Die Wissenschaftlerinnen und Wissenschaftler hoffen, dass man in Zukunft das Ausmaß der Zellschädigung bei einer Coronavirus-Infektion anhand der Gensignatur voraussagen und dann mit Medikamenten, die im Zellkern angreifen, die weitere Aktivierung dieser Gene verhindern kann.
Prof. Kracht hebt die intensive Zusammenarbeit der beteiligten Wissenschaftlerinnen und Wissenschaftler hevor: „Nur die von der Deutschen Forschungsgemeinschaft geförderten Forschungsverbünde TRR81 ‚Chromatin changes in differentiation and malignancies‘ und SFB1021 ‚RNA viruses: RNA metabolism, host response and pathogenesis‘ bündeln die für ein derartig aufwändiges Projekt notwendige biomedizinische Expertise, stellen die Plattformen an wissenschaftlichen Methoden bereit und ermöglichen uns so eine internationale Sichtbarkeit in diesem Forschungsbiet.“ (idw, red)
Literatur:
Poppe M, Wittig S, Jurida L, et al. 2017: The NF-κB-dependent and -independent transcriptome and chromatin landscapes of human coronavirus 229E-infected cells. PLoS Pathog 13:e1006286. DOI: 10.1371/journal.ppat.1006286.
Artikel teilen