Forscher der Universität Lübeck und des Instituts Pasteur in Paris haben erstmalig einen molekularen Schalter für die virale Vervielfältigung entdeckt. In Zusammenarbeit haben sie den Schalter in der Familie der Flaviviren gefunden. Mithilfe struktureller und molekularbiologischer Methoden wurde die detaillierte Untersuchung der Regulation möglich.
Viraler Lebenszyklus
Dabei ist das erforschte Feld wirtschaftlich hoch relevant. Pestiviren, wie der Virus der klassischen Schweinepest, oder auch humanpathogene Viren, wie das Hepatitis-C-Virus oder das Dengue-Virus, sind aufgrund ihrer Verbreitung und Auswirkungen ökonomisch bedeutungsvoll. Während des Zusammenbaus (Assemblierung) sind bei diesen Viren verschiedene zelluläre Komponenten, Strukturproteine und Nichtstrukturproteine (NS) involviert. Die Nichtstrukturproteine bilden dazu die viralen Replikationskomplexe aus.
Um die zeitliche Abfolge der einzelnen Stadien des viralen Lebenszyklus zu gewährleisten, werden diese Prozesse exakt reguliert. Die Regulation selbst basiert auf der Assemblierung verschiedener viraler Subkomplexe mit unterschiedlichen Funktionen. Sie bestimmen entweder die Genomreplikation oder die sich daran anschließende Bildung der Viruspartikel.
Effektivität der Viren
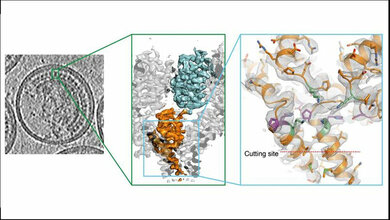
In der Studie konnte nun eine Struktur des funktionellen NS3/4A-Komplexes ermittelt werden, die detailliert die Oberflächeninteraktion zwischen den beiden Proteinen zeigt. Der Kontakt zwischen NS3 und der NS4A-kink Region ist essentiell für die virale RNA-Replikation. Eine offene räumliche Anordnung (Konformation) ist hingegen Voraussetzung für die Bildung einzelner infektiöser Viruspartikel außerhalb einer Zelle (Virionen).
„Wir zeigen in dieser Arbeit, dass das multifunktionelle NS3-Protein alternative Protein-Kontakte mit NS4A innerhalb verschiedener Subkomplexe eingeht und dadurch ein Umschalten von der Genomreplikation zur Bildung infektiöser Viruspartikel ausgelöst werden kann", erläutert Prof. Norbert Tautz vom Institut für Virologie und Zellbiologie der Universität zu Lübeck. „Unsere Ergebnisse sind ein exzellentes Beispiel dafür, wie Viren ihr begrenztes Proteinrepertoire effektiv nutzen, um eine geordnete Abfolge verschiedener Stadien des viralen Lebenszyklus zu gewährleisten." (idw, red)
Danilo Dubrau, M. Alejandra Tortorici, Félix A. Rey, Norbert Tautz: A positive-strand RNA virus uses alternative protein-protein interactions within a viral protease/cofactor complex to switch between RNA replication and virion morphogenesis. PLoS Pathog. 2017 Feb 2; 13 (2): e1006134. DOI: 10.1371/journal.ppat.1006134.
Artikel teilen