Chorea Huntington: Neue Erkenntnisse gewonnen
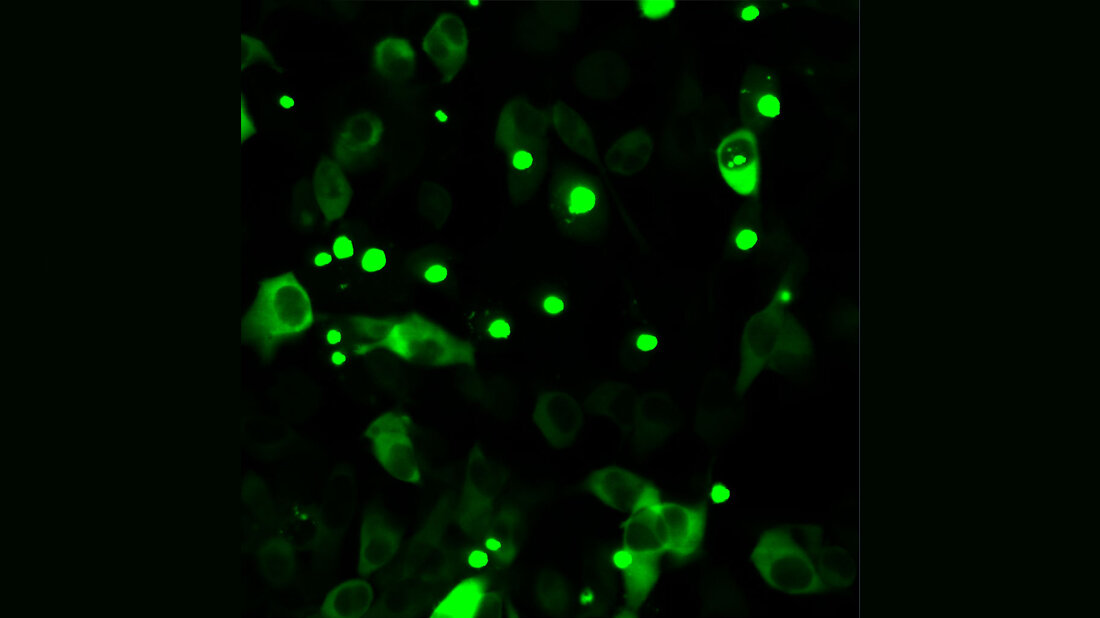
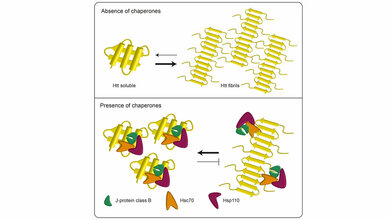
Chorea Huntington ist eine Erbkrankheit, die durch eine Mutation im Protein Huntingtin verursacht wird. Sie ist nicht therapierbar, führt zu kognitiven und motorischen Beeinträchtigungen und schließlich zum Tod. Wissenschaftler/-innen der Universität Bremen haben mit internationalen Partnern nun neue Erkenntnisse gewonnen. „Wir haben einen Mechanismus aufgeklärt, mit dem sogenannte körpereigene Proteinfaltungshelfer das mutierte Huntingtinprotein in Schach halten“, erklärt die Projektleiterin und Professorin Janine Kirstein der Universität Bremen. Proteinfaltungshelfer ermöglichen, dass Proteine ihre richtige Struktur einnehmen und bewahren, um ihre vielfältigen Funktionen ausüben zu können. Drei der Proteinfaltungshelfer kannten die Forscherinnen und Forscher bereits. Was sie aber noch nicht wussten: Wie sieht die Bindung mit dem mutierten Huntingtinprotein genau aus? Welches der drei Faltungshelfer erkennt das mutierte Protein und wie sieht die Bindung aus? „Dies konnten wir nun mit der Methode Crosslinking Massenspektrometrie identifizieren“, sagt die Biochemikerin. Mit dem Verfahren lassen sich Proteinwechselwirkungen exakt bestimmen. Zu einem Verständnis der Bindung war es allerdings noch ein weiter Weg. „Erst durch Modellierungen konnten wir die Interaktion zwischen Proteinfaltungshelfer und mutiertem Huntingtin besser verstehen.“
Noch nicht ausreichend für Therapie
„Mit dieser Arbeit ist es uns gelungen, den Mechanismus zu verstehen, wie ein Proteinfaltungshelfer ein mutiertes krankheitsassoziiertes Protein selektiv erkennt und unschädlich macht. Dies allein ist für eine therapeutische Anwendung noch nicht ausreichend“, so Janine Kirstein. „Aber man kann auf diesen Ergebnissen aufbauen und Strategien entwickeln, diese körpereigenen Faltungshelfer gezielt zu induzieren oder zu stabilisieren, um so die Toxizität von mutiertem Huntingtin zu unterdrücken.“
Erfolg durch Zusammenarbeit
„Dass wir unsere Ergebnisse so präzise gewinnen konnten, lag vor allem an der sehr guten Kooperation zwischen den Fachbereichen der Biologie/Chemie und der Produktionstechnik der Universität Bremen“, sagt Janine Kirstein „In der Biochemie brauchten wir für unser Projekt Forschende, die uns in unserer experimentellen Laborarbeit mit computergestützten Modellen unterstützen können.“ Janine Kirsteins Doktorandin Yasmin Richter fand in den Ingenieurwissenschaften in ihrer ehemaligen Kommilitonin des Masterstudiengangs Biochemistry & Molecular Biology, Isabell Grothaus, die nötige Expertise. Isabell Grothaus promoviert in der Arbeitsgruppe von Dr. Susan Köppen und Professor Lucio Colombi Ciacchi. So entstand durch die beiden Nachwuchswissenschaftlerinnen eine Kooperation zwischen den beiden Fachbereichen. „Die Ingenieurinnen haben für uns die Bindung zwischen den Proteinfaltungshelfern und dem mutierten Huntingtinprotein am Computer simuliert, und wir konnten die Modellierungen dann wiederum in unserem Labor experimentell mit gereinigten Proteinen und in Zellkulturen validieren“, erläutert Janine Kirstein.
Eine weitere Hürde war die bisher unbekannte Struktur des mutierten Huntingtinproteins. Hier konnten die Kooperationspartner Martin Kulke und Josh Vermaas der Michigan State University in den USA aushelfen, die eine Struktur postulierten, mit dem die Modellierungen am Computer durchgeführt werden konnten. Ein weiterer wichtiger Kooperationspartner war Fan Liu für die massenspektrometrischen Experimente am Leibniz-Forschungsinstitut für Molekulare Pharmakologie in Berlin, an dem Janine Kirstein vor ihrem Ruf an die Universität Bremen bis 2019 Arbeitsgruppenleiterin war.
Quelle: idw/Uni Bremen
Artikel teilen